slap in the face to epigenetics, the experiment describes what happens with some indicators of "gene expression" of human chromosome 21 in mouse hepatocytes; that is, human c21 has been incorporated into the genome of an experimental line of mice. They find that things such as the binding site of several transcription factors, and the general levels of transcription on the chromosome, are for the most (but not entirely) the same as in human hepatocytes. Their conclusion? The sequence of the human C21, rather than the epigenetic cell environment, is mostly responsible for "regulation" of its own expression. Hence, "It's the sequence, stupid".
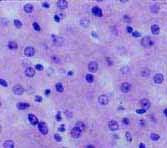
slap in the face to epigenetics, the experiment describes what happens with some indicators of "gene expression" of human chromosome 21 in mouse hepatocytes; that is, human c21 has been incorporated into the genome of an experimental line of mice. They find that things such as the binding site of several transcription factors, and the general levels of transcription on the chromosome, are for the most (but not entirely) the same as in human hepatocytes. Their conclusion? The sequence of the human C21, rather than the epigenetic cell environment, is mostly responsible for "regulation" of its own expression. Hence, "It's the sequence, stupid".But, is it? It is well-known that all cells within the body contain the same sequence, but express very differently in different cell types, because of the different cell environments. It is interesting that the people at Science managed to wash this fact out of their brains, since it directly refutes that expression is determined in the sequence. Further, their conclusion is simply not logical because they are comparing cells with the exact same cell phenotype. The image shows micrographies from two liver sections; one is from mouse, the other is from human. Can anyone tell me which is which? The fact is, at the cell-histological level, most homologous tissues of mouse and human are undistinguishable.
Don't expect virtually identical cells to produce great differences in the expression of the same sequence just because you are comparing them in different species. Expect them to produce virtually identical gene expression. As simple as that.
Refs:
2008 Genetics.It's the sequence, stupid! Science. 322:380-1.
UPDATE
Check out this site on hepatocyte histology
Left is pig; right is raccoon
 Phenotypic plasticity: This is mouse, fasted and glycogen-enriched
Phenotypic plasticity: This is mouse, fasted and glycogen-enriched And of course, human (from another site):
And of course, human (from another site):